Nieuw inzicht in hoe cellen de activiteit van hun genen regelen
RNA moleculen bevatten informatie voor het maken van eiwitten. Daarnaast kunnen ze ook een heel specifieke 2D en 3D structuur aannemen. Soms kan één RNA molecuul schakelen tussen een UIT en AAN stand, waarmee ze bepalen of het RNA zich wel of niet kan binden aan ribosomen, die de RNA code in een eiwit vertalen. Een nieuwe publicatie, onder leiding van moleculair bioloog Danny Incarnato van de RUG en geschreven door postdoc onderzoeker Dr. Ivana Borovska, is op 25 juli gepubliceerd in Nature Biotechnology en beschrijft honderden van dit soort RNA schakelaars in de E.coli bacterie en in menselijke cellen.
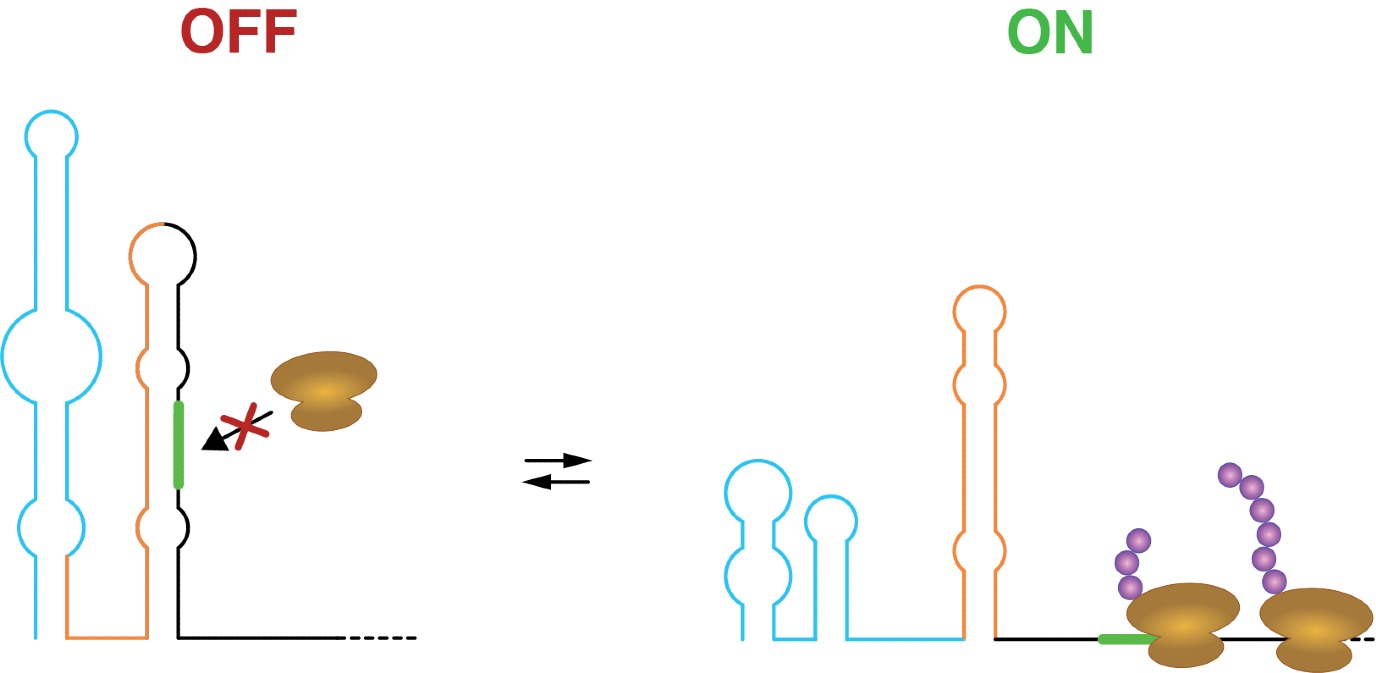
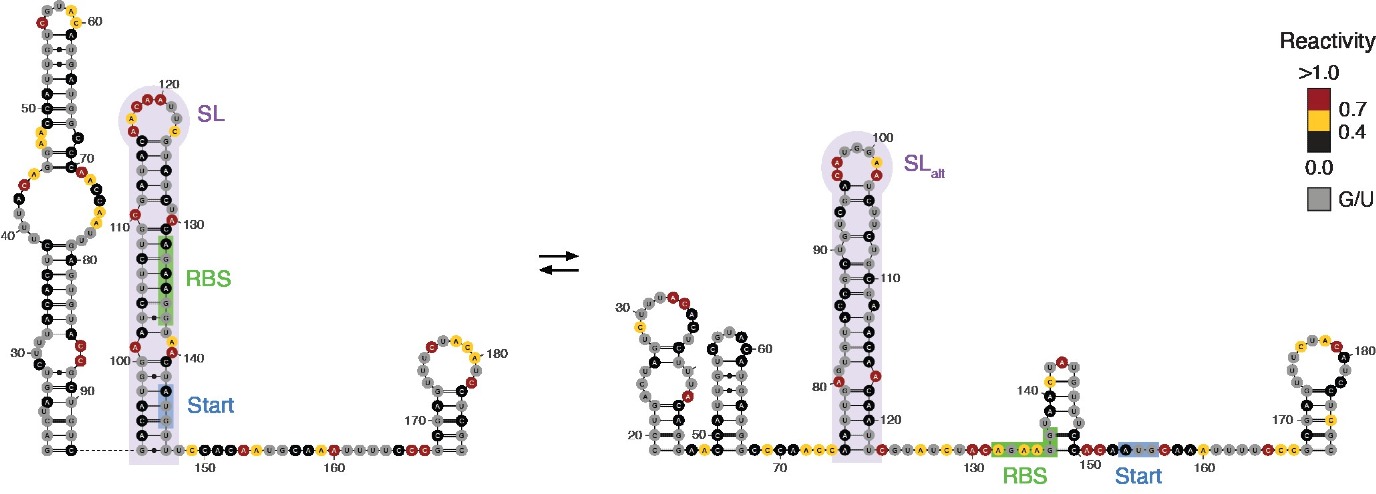
Een paar jaar geleden heeft Incarnato een methode ontwikkeld om de alternatieve structuren van RNA moleculen in levende cellen aan te tonen. Met deze methode ontdekte hij regio’s in het RNA die zorgen voor een overgang tussen twee verschillende vormen, elk met een verschillende functie. Incarnato: ‘Wanneer RNA moleculen in twee alternatieve vormen kunnen overgaan wijst dat op een regulerende functie, zoals een AAN-UIT schakelaar.’
Nieuwe behandelingen
Het team van Incarnato heeft deze methode nu gebruikt om de complexiteit van RNA structuren in levende cellen te bestuderen. Bovendien bouwden ze een model waarmee ze, met gebruik van evolutionaire informatie, kunnen identificeren welke RNA schakelaars ook echt een functie hebben. Ze vonden zo honderden functionele schakelaars. Een voorbeeld is een schakelaar die gevoelig is voor temperatuur, en die bacteriën helpt om koude-stress tegen te gaan. ‘De identificatie van een groot aantal schakelaars is een eerste stap. De volgende stap is om uit te vinden hoe we de werking kunnen beïnvloeden.’ Dat zou het bijvoorbeeld mogelijk maken om kleine moleculen te ontwerpen die het effect van de schakelaars kunnen veranderen, wat op termijn kan leiden tot nieuwe behandelingen voor ziekten.
De studie die nu gepubliceerd is duurde drie jaar, en bouwde voort op zeker zes jaar fundamenteel onderzoek naar de detectie van 2D vormen in RNA. Incarnato: ‘Dit is iets dat het onderzoeksveld kan transformeren, en waar iedereen al jaren naar op zoek was.’
Referentie: Ivana Borovská, Danny Incarnato et al. Identification of conserved RNA regulatory switches in living cells using RNA secondary structure ensemble mapping and covariation analysis. Nature Biotechnology, 25 July 2025.
Meer nieuws
-
29 januari 2026
Microplasticonderzoek - opgeblazen nieuws of een echt gevaar?
-
27 januari 2026
ERC Proof of Concept grant voor Maria Loi
